Логистическая регрессия в R
Логистическая регрессия - статистическая модель, которая применяется для предсказания вероятности возникновения некоторого события по значениям множества признаков.
Модель
Обозначим предсказываемую зависимую переменную как $y$. Она может принимать два значения $0$ - событие не произошло и $1$ - событие произошло.
Вероятность того, что событие произойдет $P(y=1)$. Тогда $P(y=0)=1 - P(y=1)$ вероятность того что событие не произойдет. Используя для предсказания независимые переменные $x_1, x_2…,x_k$ получим функцию предсказания:
$$P(y=1)=\frac{1}{1+e^{-(B_0 + B_1 * x_1+B_2 * x_2+…+B_k * x_k)}}$$
Функция возвращает вероятность того, что $y=1$, т.е. событие произошло и имеет вид:
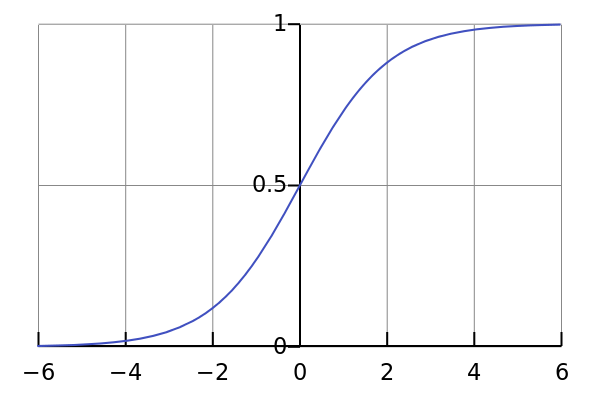
Логистическая регрессия в R
Построим модель предсказывающую вероятность выживания каждого пассажира на Титанике используя реальные данные (тренировочные, тестовые):
# Загружаем данные в dataframe
setwd("path/to/working/directory")
data <- read.csv("train.csv")
# Рассмотрим структуру данных
str(quality)
#'data.frame': 891 obs. of 12 variables:
# $ PassengerId: int 1 2 3 4 5 6 7 8 9 10 ...
# $ Survived : int 0 1 1 1 0 0 0 0 1 1 ...
# $ Pclass : int 3 1 3 1 3 3 1 3 3 2 ...
# $ Name : Factor w/ 891 levels "Abbing, Mr. Anthony",..: 109 191 358 277 16 559 520 629 417 581 ...
# $ Sex : Factor w/ 2 levels "female","male": 2 1 1 1 2 2 2 2 1 1 ...
# $ Age : num 22 38 26 35 35 NA 54 2 27 14 ...
# $ SibSp : int 1 1 0 1 0 0 0 3 0 1 ...
# $ Parch : int 0 0 0 0 0 0 0 1 2 0 ...
# $ Ticket : Factor w/ 681 levels "110152","110413",..: 524 597 670 50 473 276 86 396 345 133 ...
# $ Fare : num 7.25 71.28 7.92 53.1 8.05 ...
# $ Cabin : Factor w/ 148 levels "","A10","A14",..: 1 83 1 57 1 1 131 1 1 1 ...
# $ Embarked : Factor w/ 4 levels "","C","Q","S": 4 2 4 4 4 3 4 4 4 2 ...
# Немного очистим данные
# Заменим строки, в которых отсутствует возраст медианой
data$Age[is.na(data$Age)] <- median(data$Age, na.rm=TRUE)
# Создадим модель зависимости выживания пассажира от его пола,
# класса которым он путешествует и возраста.
# family = binomial именно этот параметр указывает что мы хотим использовать логистическую регрессию
model = glm(Survived ~ Sex + Pclass + Age, data=data, family = binomial)
# Рассмотрим нашу модель
summary(QualityLog)
# Estimate указывает коэффициент Bi перед независимой переменной
# Количество "*" указывает на значимость переменной в модели,
# в нашем случае все независимые влияют на зависимую очень сильно
#Coefficients:
# Estimate Std. Error z value Pr(>|z|)
# (Intercept) 4.724739 0.449822 10.504 < 2e-16 ***
# Sexmale -2.612279 0.186505 -14.006 < 2e-16 ***
# Pclass -1.171719 0.119318 -9.820 < 2e-16 ***
# Age -0.033311 0.007371 -4.519 6.2e-06 ***
# ---
# Signif. codes: 0 '**' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
#(Dispersion parameter for binomial family taken to be 1)
#Null deviance: 1186.66 on 890 degrees of freedom
#Residual deviance: 805.59 on 887 degrees of freedom
#AIC: 813.59
# Загрузим тестовые данные
test <- read.csv("test.csv")
# Используем модель для предсказаний на тестовых данных
# predictResult содержит вектор с вероятностью выживания для каждого пассажира
predictResult <- predict(model, newdata = test, type="response")
# Теперь в тестовой выборке значение Survived установлено в 1
# если вероятность положительного исхода больше либо равна 50%
# и 0 в противном случае
test$Survived[predictResult >= 0.5] = 1
test$Survived[predictResult < 0.5] = 0
# На реальных данных модель правильно предсказывает результат в 74.641% случаев.
Receiver Operator Characteristics (ROC) кривая
Результатом логистической регрессии является вероятность того что событие произойдет, но чтобы сделать предсказание нам необходимо определить пороговое (threshold) значение $t$, такое что:
- $P(y = 1) >= t$, событие произошло
- $P(y = 1) < t$, событие не произошло
При высоком пороговом значении модель редко будет предсказывать положительный результат (только при высокой вероятности $P(y=1)$). И обратно, при низком пороговом значении модель будет чаще предсказывать положительный исход и реже отрицательный.
Для численного представления качества оценки используют матрицу неточностей (confusion matrix).
| Predicted = 0 | Predicted = 1 | |
|---|---|---|
| Actual = 0 | True Negatives (TN) | False Positives (FP) |
| Actual = 1 | False Negatives (FN) | True Positives (TP) |
В таблице мы видим как часто наша модель делает правильные предсказания:
- TN - предсказано False, в действительности False
- TP - предсказано True, в действительности True
И как часто ошибается:
- FP - предсказано True, в действительности False
- FN - предсказано False, в действительности True
Отсюда можно получить два значения, которые помогут нам определить какие ошибки делает модель:
$Sensitivity = TP / (TP + FN)$ - чувствительность, или True positive rate. Процент верно предсказанных позитивных исходов.
$Specificity = TN / (TN + FP)$ - специфичность или True negative rate. Процент верно предсказанных негативных исходов.
Модель с более высоким пороговым значением будет иметь более высокую чувствительность и низкую специфичность. Модель с низким пороговым значением наоборот.
Если предпочтений нет можно оставить пороговое значение $t = 0.5$.
# Используем модель для предсказания на тестовых данных
# для этого не указываем параметр newdata
predictTrain <- predict(model, type="response")
# 0.5 в данном случае является пороговым значением
table(data$Survived, predictTrain > 0.5)
# Модель верно предсказывает отрицательный исход в 454 случаях из 594
# И положительный исход в 294 случаях из 342
# FALSE TRUE
# 0 454 95
# 1 93 249
sensitivity <- 249 / (93 + 249) # 0.7280702
specificity <- 454 / (454 + 95) # 0.8269581
Подобрать необходимое пороговое значение можно с помощью ROC-кривой.
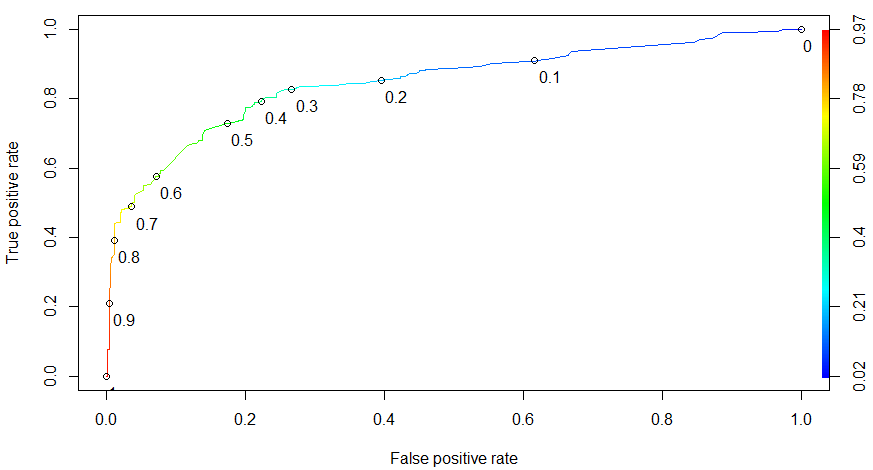
True negative rate указывается на оси абсцисс, True positive rate на оси ординат. Кривая показывает соотношения этих величин при разных значения пороговой величины.
Построить ROC-кривую в R можно следующим набором команд:
# Устанавливаем необходимый пакет
install.package("ROCR")
package(ROCR)
# Строим ROC-кривую
ROCRpred = prediction(predictTrain, data$Survived)
ROCRperf = performance(ROCRpred, "tpr", "fpr")
plot(ROCRperf, colorize=TRUE, print.cutoffs.at=seq(0,1,0.1), text.adj=c(-0.2,1.7))